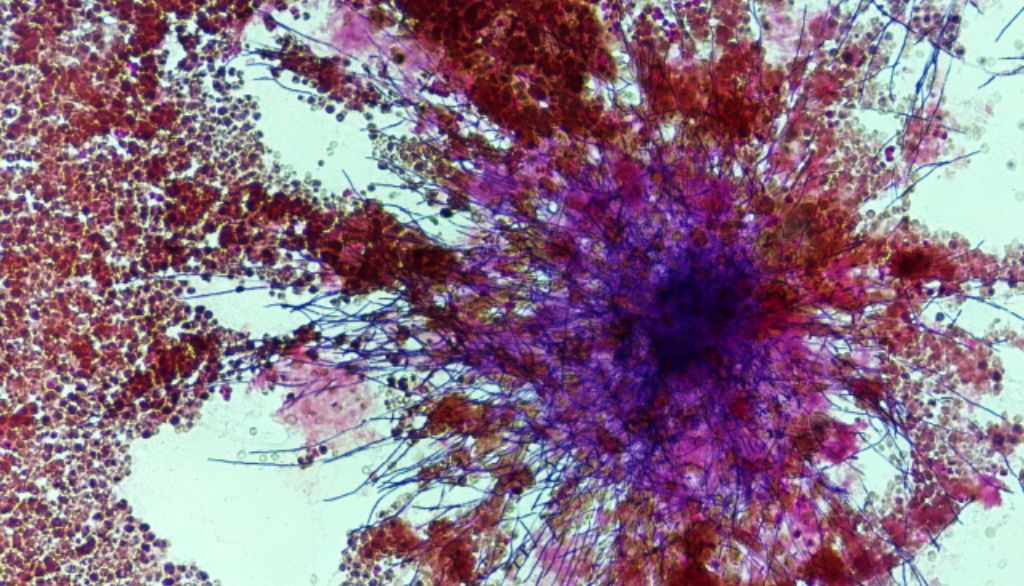
Candida albicans est un champignon qui cause des infections généralement bénignes. Par contre, chez les personnes immunosupprimées, il peut causer des infections systémiques graves, voire des décès. Des souches résistantes aux antibiotiques ont fait leur apparition chez cette espèce.
— Antonio e Biagio e Cesare Arrigo
Une étude publiée dans Nature Ecology and Evolution par des chercheurs de l'Université Laval suggère une façon de déterminer rapidement si une souche de champignon pathogène est résistante à un antibiotique donné. Grâce à l'approche proposée par ces chercheurs, les délais pour choisir un traitement efficace pourraient être réduits de moitié.
«Chez les personnes en bonne santé, les infections causées par les champignons sont généralement bénignes», souligne le responsable de l'étude, Christian Landry, professeur à la Faculté des sciences et de génie de l'Université Laval et chercheur à l'Institut de biologie intégrative et des systèmes ainsi qu'au regroupement PROTEO. «Il en va autrement chez les personnes dont le système immunitaire est fortement affaibli. Chez les personnes immunosupprimées, une simple mycose peut avoir des répercussions graves, voire mortelles. Il est donc important de choisir le bon traitement et de leur administrer sans délai.»
Présentement, il faut compter de 1 à 2 semaines pour franchir toutes les étapes menant à la sélection d'un antifongique adéquat. Il faut prélever un échantillon contenant le champignon pathogène, il faut ensuite le cultiver en laboratoire et, finalement, il faut tester différents antifongiques pour éviter de prescrire un produit pour lequel cette souche possède une résistance. «Il existe moins de cinq classes d'antifongiques contre les champignons pathogènes chez l'humain, et plusieurs espèces de champignons possèdent des gènes de résistance contre toutes ces classes», précise le professeur Landry.
Pour accélérer le processus, le chercheur et son équipe proposent de créer des catalogues de mutations qui permettraient de déterminer, à partir du séquençage du génome d'un champignon, s'il est résistant à un antifongique donné. «Notre approche consiste à créer des variants qui n'ont pas encore été vus en milieu clinique, mais qui pourraient l'être éventuellement. Si cela se produisait, nous saurions rapidement quel antifongique il faut éviter», explique le professeur Landry.
Afin de faire une preuve de concept, les chercheurs ont créé, à l'aide de l'outil d'édition génomique CRISPR-Cas, environ 3000 variants d'une même protéine, la cytosine déaminase, impliquée dans la résistance à un antifongique, la fluorocytosine. Par la suite, ces variants, qui ne se distinguaient les uns des autres que par un seul acide aminé, ont été insérés dans des levures de boulanger. L'exercice a permis d'établir que 1091 de ces variants conféraient une résistance à la fluorocytosine. C'est quatre fois plus que le nombre de variants résistants aux antifongiques qui avaient été répertoriés jusqu'à présent.
— Christian Landry
«Il faudrait créer des catalogues comme celui-là pour la vingtaine de protéines impliquées dans la résistance aux antibiotiques chez les champignons, suggère Christian Landry. Avec ces outils, nous devrions être en mesure de répondre plus rapidement et plus adéquatement lorsque de nouveaux variants feront leur apparition en milieu clinique.»
Les signataires de l'étude parue dans Nature Ecology and Evolution sont Philippe Després, Angel Cisneros, Emilie Alexander, Ria Sonigara, Cynthia Gagné-Thivierge, Alexandre Dubé et Christian Landry.